페니실린의 발견 이후 항생제는 감염병 치료에 혁신을 가져왔지만, 오남용이 이어지면서 여러 약물에 동시에 내성을 갖는 다제내성균(MDR)*이 유전자 변이를 통해 등장해 인류 건강에 새로운 위협이 되고 있다.
 이런 상황에서 국내 연구진이 내성균에 특화된 신약 후보를 신속히 발굴할 수 있는 인공지능(AI) 기반의 차세대 항생제 개발 기술을 선보여 주목받고 있다.
이런 상황에서 국내 연구진이 내성균에 특화된 신약 후보를 신속히 발굴할 수 있는 인공지능(AI) 기반의 차세대 항생제 개발 기술을 선보여 주목받고 있다.
▲(왼쪽부터) 남호정 교수 서지원 교수 배대훈 석사 김민상 박사과정생
광주과학기술원(GIST, 총장 임기철)은 전기전자컴퓨터공학과 남호정 교수와 화학과 서지원 교수 공동연구팀이 다양한 박테리아의 유전자 정보와 항균 펩타이드 간의 활성 관계를 분석해, 균종 특이적인 펩타이드 기반 항생제 후보물질을 제안하는 AI 모델을 개발했다고 밝혔다.
이 모델은 박테리아 종별 고유 유전자 정보와 다양한 항균 펩타이드 간의 상관관계 데이터를 학습하여 감염병을 유발한 세균에 최적화된 항균 펩타이드를 선별할 수 있다. 이를 통해 균종 특이적 정밀의료는 물론, 유전자 변이를 거쳐 기존 항생제에 내성을 지닌 병원균에도 정밀하게 대응할 수 있는 맞춤형 치료제 후보 도출이 가능해졌다.
지금까지의 AI 기반 항균 펩타이드 연구는 단순히 항균 활성 여부만을 예측하거나, 표적 박테리아 종을 고려하지 않아 실제 활용에 한계가 있었다.
연구팀은 이를 극복하기 위해 대용량 펩타이드 데이터를 학습시키고, 표적 박테리아 종의 게놈 정보를 활용하는 AI 모델인 ‘램프(LLAMP·Large Language model for AMP activity prediction)’를 세계 최초로 개발했다.
‘램프’는 특정 박테리아의 게놈 정보와 펩타이드 서열을 입력하면, 해당 박테리아 종에 대한 펩타이드의 활성지표로서 최소억제농도(MIC, Minimum Inhibitory Concentration)*를 예측한다.
이 모델은 단백질 데이터를 기반으로 사전 학습된 언어 모델에 대용량 펩타이드 데이터를 추가로 학습시켜 펩타이드 특유의 ‘언어’를 이해하도록 만든 뒤, 박테리아 게놈–펩타이드 조합을 기반으로 미세 조정(Fine-tuning)*을 거쳐 완성됐다.
기존 모델보다 항균성 예측 정확도가 최소 4%, 최대 9% 향상되고, 활성값 예측력은 최소 3%에서 최대 40%까지 개선되는 등 모든 성능 지표에서 우수한 성능을 보였다.
특히 연구팀은 ‘램프’를 통해 자연계에 존재하는 약 500만 개의 펩타이드 서열을 스크리닝(screening)하고, 어텐션(Attention) 분석*을 통해 항균 활성에 기여하는 특정 아미노산들을 확인했다.
이 분석 결과를 바탕으로 펩타이드를 재설계(서열 엔지니어링)하여 항균 특성을 더욱 높였고, 병원성 세균에 직접 적용해 항균 활성을 확인했다.
그 결과, 병원균의 유전자 정보를 입력해 스크리닝*한 후보물질들이 강한 독성과 내성을 지닌 병원균(ESKAPE*)에 대해서도 최대 3.1 마이크로몰(μM)의 최소억제농도값(MIC)을 나타내며 강력한 항균 효과를 보였다. 이를 통해 실제 치료제 후보로서의 가능성이 높다는 것을 실험적으로 입증했다.
아울러, 이들 펩타이드 화합물을 대상으로 적혈구에 대한 용혈독성* 시험을 진행한 결과, 실제 임상 3상까지 진행된 펩타이드 항생제 ‘펙시가난(pexiganan)*’과 비슷한 수준의 안전성(용혈독성)과 선택성을 보였다.
또한 추가 연구를 통해, 활성과 선택성이 우수한 항균 펩타이드가 나선형 구조에서 유래한 특정 아미노산의 배열과 양친매성* 특성을 통해 세균의 세포막을 직접 파괴하는 메커니즘으로 작용한다는 사실도 규명했다.
연구팀은 이번 성과에 대해 AI가 단순히 기존 약물을 모방하는 데 그치지 않고, 병원균의 유전자 특성을 분석해 그에 최적화된 새로운 치료제를 설계할 수 있음을 보여준 데에 큰 의미가 있다고 평가했다.
특히 항생제 내성의 진화를 실시간으로 추적·대응할 수 있는 신약개발 플랫폼을 제시한 성과로, 학술적·산업적 파급력이 매우 크다는 점에서 주목된다.
남호정 교수는 “새로운 내성균이 등장했을 때, 그 유전자 정보를 기반으로 빠르게 항생제 후보물질을 제안할 수 있는 AI 기반 신약개발 체계를 구축한 것이 이번 연구의 핵심”이라며, “균종 특이적 펩타이드를 발굴해 내성균에 특화된 항생제를 개발할 수 있다는 점에서 기존 모델과 차별화된다”고 밝혔다.
GIST 전기전자컴퓨터공학과 남호정 교수와 화학과 서지원 교수가 지도하고 전기전자컴퓨터공학과 배대훈 석사, 화학과 김민상 박사과정생이 수행한 이번 연구는 과학기술정보통신부·한국연구재단의 중견연구자지원사업, 지역혁신 선도연구센터(RLRC), 바이오의료기술개발사업 프로그램과 보건복지부·과학기술정보통신부의 연합학습 기반 신약개발 가속화 프로젝트(K-MELLODDY)의 지원을 받았다. 연구 결과는 국제학술지 《브리핑스 인 바이오인포매틱스(Briefings in Bioinformatics)》에 2025년 7월 18일 온라인으로 게재됐다.
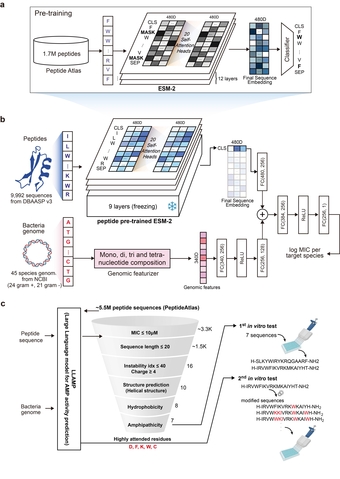
▲[그림1] LLAMP 모델 개요. a) 사전학습 과정: LLAMP 모델은 약 170만 개의 펩타이 서열로 사전학습되어 서열 패턴을 학습함. b) 미세조정 과정: 이후 균종별 활성을 학습해 MIC 값을 예측함. c) AI 기반 스크리닝 과정: 학습된 모델과 펩타이드 특성을 활용해 효과적인 후보를 선별함.
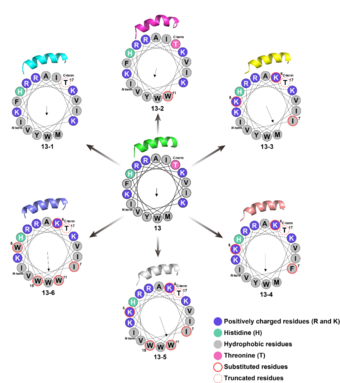
▲[그림2] 펩타이드 13 및 유도체의 구조 분석. 나선휠과 3D 구조를 통해, 양전하(보라색), 소수성(회색) 등 잔기의 특성과 치환/절단 부위를 시각화함. 화살표는 소수성 모멘트를, ‘N-term‘, ‘C-term‘ 표시는 말단 위치를 나타냄.












